Medidas de Dispersión#
Última modificación: Mar 11, 2024 | YouTube
[1]:
import warnings
warnings.filterwarnings("ignore")
[2]:
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import seaborn as sns
Definición del problema#
El conjunto de datos de la flor Iris contiene 150 muestras de las medidas del ancho y el largo del pétalo y del sépalo para las tres especies de esta flor (Iris setosa, Iris virginica e Iris versicolor). A partir de la muestra de datos se desean responder la siguiente pregunta:
P2.— ¿Cuál es el rango típico de la longitud y ancho del pétalo y del sépalo para cada uno de los tipos de flores?
Carga de datos#
[3]:
#
# Se cargan los datos y se realiza una inspección
# inicial de la tabla y los datos
#
iris = sns.load_dataset("iris")
display(iris.head(), iris.tail())
| sepal_length | sepal_width | petal_length | petal_width | species | |
|---|---|---|---|---|---|
| 0 | 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 1 | 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 2 | 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 3 | 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 4 | 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| sepal_length | sepal_width | petal_length | petal_width | species | |
|---|---|---|---|---|---|
| 145 | 6.7 | 3.0 | 5.2 | 2.3 | virginica |
| 146 | 6.3 | 2.5 | 5.0 | 1.9 | virginica |
| 147 | 6.5 | 3.0 | 5.2 | 2.0 | virginica |
| 148 | 6.2 | 3.4 | 5.4 | 2.3 | virginica |
| 149 | 5.9 | 3.0 | 5.1 | 1.8 | virginica |
P2.— ¿Cuál es el rango típico de la longitud y ancho del pétalo y del sépalo para cada uno de los tipos de flores?#
Esta pregunta se refiere a una medida de la dispersión o variabilildad de los datos y a como se distribuyen.
Métricas basadas en la distancia respecto a las métricas de localización#
Desviación:
Diferencia entre el valor observado y el valor estimado de localización. En modelado sus sinónimos son errores o residuales.
Rango:
Diferencia entre el valor más grande y más pequeño de la muestra de los datos.
[4]:
iris_melt = pd.melt(
iris,
id_vars="species",
var_name="Variables",
value_name="Values",
)
[5]:
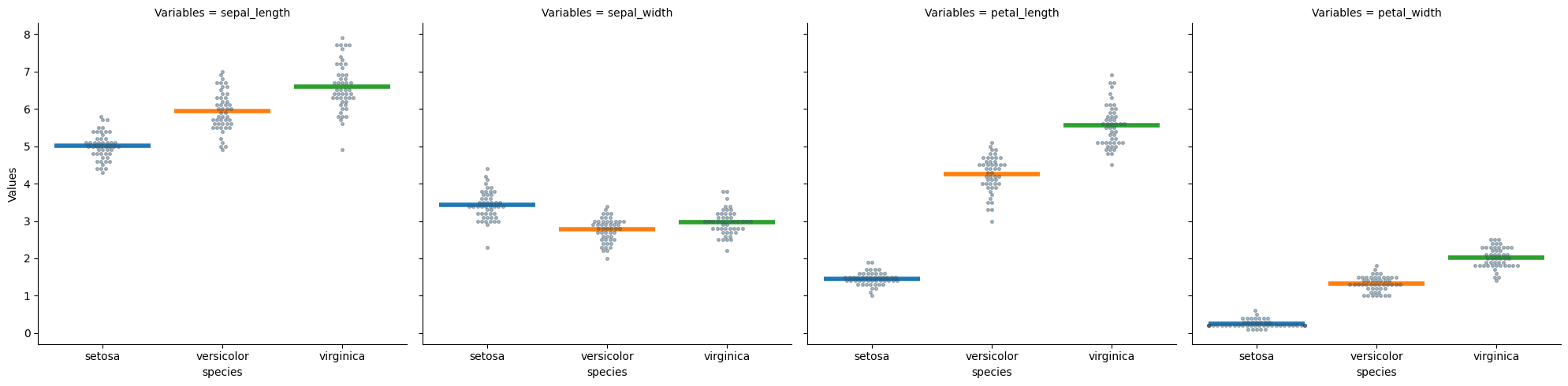
g = sns.catplot(
x="species",
y="Values",
data=iris_melt,
col="Variables",
kind="swarm",
col_wrap=4,
alpha=0.4,
s=7,
edgecolors="k",
linewidth=1,
)
colors = ["tab:blue", "tab:orange", "tab:green"]
means = iris.groupby("species").mean()
for i_axes in range(4):
ax = g.axes[i_axes]
x_lim = ax.get_xlim()
mean_values = means[means.columns[i_axes]]
for i, (color, mean_value) in enumerate(zip(colors, mean_values)):
g.axes[i_axes].hlines(mean_value, i - 0.4, i + 0.4, ls="-", color=color, lw=4)
plt.show()
Varianza muestral:
s^2 = \frac{1}{n-1} \sum_{i=1}^n (x_i - \bar{x})^2
[6]:
#
# Cálculo para una variable
#
from statistics import variance
display(
iris.sepal_length.var(),
variance(iris.sepal_length),
np.var(iris.sepal_length),
)
0.6856935123042507
0.6856935123042506
0.6811222222222223
[7]:
#
# Cálculo para las columnas del dataframe
#
iris.var(numeric_only=True)
[7]:
sepal_length 0.685694
sepal_width 0.189979
petal_length 3.116278
petal_width 0.581006
dtype: float64
[8]:
#
# Cálculo por especie
#
iris.groupby("species").var()
[8]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 0.124249 | 0.143690 | 0.030159 | 0.011106 |
| versicolor | 0.266433 | 0.098469 | 0.220816 | 0.039106 |
| virginica | 0.404343 | 0.104004 | 0.304588 | 0.075433 |
Desviación estándar muestral.
Raíz cuadrada de la varianza muestral.
[9]:
#
# Cálculo para una variable
#
from statistics import stdev
display(
iris.sepal_length.std(),
stdev(iris.sepal_length),
np.std(iris.sepal_length),
)
0.828066127977863
0.8280661279778629
0.8253012917851409
[10]:
#
# Cálculo para las columnas del dataframe
#
iris.std(numeric_only=True)
[10]:
sepal_length 0.828066
sepal_width 0.435866
petal_length 1.765298
petal_width 0.762238
dtype: float64
[11]:
#
# Cálculo por especie
#
iris.groupby("species").std()
[11]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 0.352490 | 0.379064 | 0.173664 | 0.105386 |
| versicolor | 0.516171 | 0.313798 | 0.469911 | 0.197753 |
| virginica | 0.635880 | 0.322497 | 0.551895 | 0.274650 |
Desviación media absoluta (MAD):
Norma L1 o distancia de Manhattan.
\text{MAD} = \frac{1}{n} \sum_{i=1}^n |x_i - \bar{x}|
[12]:
#
# Cálculo para las columnas del dataframe
#
from scipy.stats import median_abs_deviation
iris[["sepal_length", "sepal_width", "petal_length", "petal_width"]].apply(
median_abs_deviation, axis=0
)
[12]:
sepal_length 0.70
sepal_width 0.30
petal_length 1.25
petal_width 0.70
dtype: float64
[13]:
#
# Cálculo por especie
#
iris.groupby("species").apply(median_abs_deviation)
[13]:
species
setosa [0.20000000000000018, 0.25, 0.1000000000000000...
versicolor [0.3500000000000001, 0.20000000000000018, 0.34...
virginica [0.40000000000000036, 0.20000000000000018, 0.4...
dtype: object
Desviación absoluta de la mediana:
Ya que la varianza y la desviación media absoluta no son robusta a outliers, se puede realizar un estimado de la desviación usando la mediana m de la muestra:
\text{Mediana}(|x_1 - m|,\; |x_2 - m|,\;...,\;|x_n - m|)
También es posible computar la varianza muestral truncada en analogía a la media truncada.
[14]:
def madm(df):
d = df._get_numeric_data().copy()
m = d.median()
for c in d.columns:
d[c] = d[c] - m[c]
return d.apply(median_abs_deviation)
madm(iris)
[14]:
sepal_length 0.70
sepal_width 0.30
petal_length 1.25
petal_width 0.70
dtype: float64
[15]:
#
# Cálculo por especie
#
def madm(x):
m = x.median()
x = x - m
x = x.abs()
return x.mean()
iris.groupby("species").agg(madm)
[15]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 0.270 | 0.284 | 0.130 | 0.066 |
| versicolor | 0.420 | 0.250 | 0.376 | 0.154 |
| virginica | 0.492 | 0.238 | 0.440 | 0.226 |
Métricas basadas en la distribución de los datos#
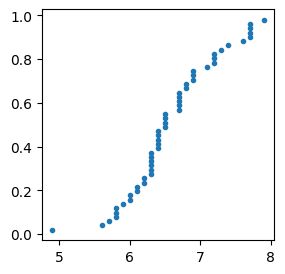
Función de Densidad Acumulada Empírica (ECDF)
[16]:
def ecdf(ax, data, param_dict=None):
if param_dict is None:
param_dict = {}
props = {
"marker": ".",
"linestyle": "none",
}
props = {**props, **param_dict}
x = np.sort(data)
y = np.arange(1, len(data) + 1) / (len(data) + 1)
out = ax.plot(x, y, **props)
return out
_, ax = plt.subplots(1, 1, figsize=(3, 3))
ecdf(ax, iris.sepal_length[iris.species == "virginica"])
plt.show()
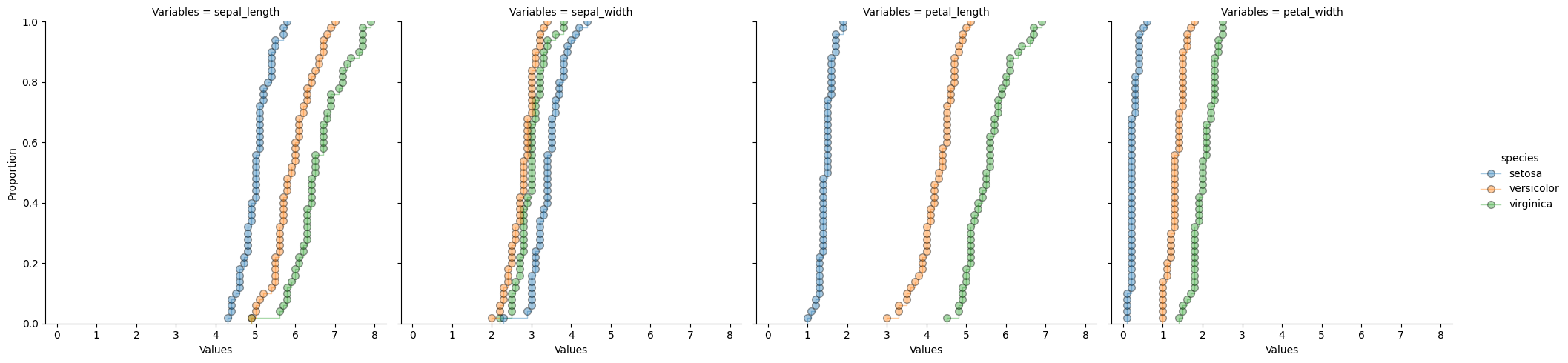
[17]:
sns.displot(
kind="ecdf",
x="Values",
hue="species",
data=iris_melt,
col="Variables",
col_wrap=4,
marker="o",
linestyle=None,
alpha=0.4,
markersize=7,
markeredgecolor="k",
linewidth=1,
)
plt.show()
Estadísticos de orden: Métricas calculadas sobre los datos ordenados.
Percentil p: Valor para el cual el p por ciento de los datos son menores o iguales.
decil d: Valor para el cual el d por ciento de los datos son menores o iguales. d es un múltiplo de 10.
Cuartil inferior: Valor para el cual el 25% de las observaciones son más pequeñas (percentil del 25%).
Cuartil superior: Valor para el cual el 25% de las observaciones son más grandes (percentil del 75%).
Rango intercuartil (IQR):
\text{IQR} = \text{Cuartil superior} - \text{Cuartil inferior}
[18]:
#
# Cálculo de los cuartiles
#
iris.quantile(
# -------------------------------------------------------------------------
# the quantile(s) to compute
q=[0.25, 0.75],
# -------------------------------------------------------------------------
# Specifies the interpolation method to use, when the desired quantile lies
# between two data points i and j:
# {‘linear’, ‘lower’, ‘higher’, ‘midpoint’, ‘nearest’}
interpolation="linear",
# -------------------------------------------------------------------------
numeric_only=True,
)
[18]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| 0.25 | 5.1 | 2.8 | 1.6 | 0.3 |
| 0.75 | 6.4 | 3.3 | 5.1 | 1.8 |
[19]:
#
# Cálculo de cuartiles
#
display(iris.quantile(0.25, numeric_only=True), iris.quantile(0.75, numeric_only=True))
sepal_length 5.1
sepal_width 2.8
petal_length 1.6
petal_width 0.3
Name: 0.25, dtype: float64
sepal_length 6.4
sepal_width 3.3
petal_length 5.1
petal_width 1.8
Name: 0.75, dtype: float64
[20]:
iris.groupby("species").quantile(0.25)
[20]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 4.800 | 3.200 | 1.4 | 0.2 |
| versicolor | 5.600 | 2.525 | 4.0 | 1.2 |
| virginica | 6.225 | 2.800 | 5.1 | 1.8 |
[21]:
iris.groupby("species").quantile(0.75)
[21]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 5.2 | 3.675 | 1.575 | 0.3 |
| versicolor | 6.3 | 3.000 | 4.600 | 1.5 |
| virginica | 6.9 | 3.175 | 5.875 | 2.3 |
[22]:
iris.groupby("species").quantile(0.75) - iris.groupby("species").quantile(0.25)
[22]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 0.400 | 0.475 | 0.175 | 0.1 |
| versicolor | 0.700 | 0.475 | 0.600 | 0.3 |
| virginica | 0.675 | 0.375 | 0.775 | 0.5 |
[23]:
#
# El método describe de pandas computa los principales estadísticos para las
# columnas numéricas del dataframe
#
iris.describe()
[23]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| count | 150.000000 | 150.000000 | 150.000000 | 150.000000 |
| mean | 5.843333 | 3.057333 | 3.758000 | 1.199333 |
| std | 0.828066 | 0.435866 | 1.765298 | 0.762238 |
| min | 4.300000 | 2.000000 | 1.000000 | 0.100000 |
| 25% | 5.100000 | 2.800000 | 1.600000 | 0.300000 |
| 50% | 5.800000 | 3.000000 | 4.350000 | 1.300000 |
| 75% | 6.400000 | 3.300000 | 5.100000 | 1.800000 |
| max | 7.900000 | 4.400000 | 6.900000 | 2.500000 |
[24]:
for column in iris._get_numeric_data().columns:
print(column)
print(iris.groupby("species")[column].describe())
print("")
sepal_length
count mean std min 25% 50% 75% max
species
setosa 50.0 5.006 0.352490 4.3 4.800 5.0 5.2 5.8
versicolor 50.0 5.936 0.516171 4.9 5.600 5.9 6.3 7.0
virginica 50.0 6.588 0.635880 4.9 6.225 6.5 6.9 7.9
sepal_width
count mean std min 25% 50% 75% max
species
setosa 50.0 3.428 0.379064 2.3 3.200 3.4 3.675 4.4
versicolor 50.0 2.770 0.313798 2.0 2.525 2.8 3.000 3.4
virginica 50.0 2.974 0.322497 2.2 2.800 3.0 3.175 3.8
petal_length
count mean std min 25% 50% 75% max
species
setosa 50.0 1.462 0.173664 1.0 1.4 1.50 1.575 1.9
versicolor 50.0 4.260 0.469911 3.0 4.0 4.35 4.600 5.1
virginica 50.0 5.552 0.551895 4.5 5.1 5.55 5.875 6.9
petal_width
count mean std min 25% 50% 75% max
species
setosa 50.0 0.246 0.105386 0.1 0.2 0.2 0.3 0.6
versicolor 50.0 1.326 0.197753 1.0 1.2 1.3 1.5 1.8
virginica 50.0 2.026 0.274650 1.4 1.8 2.0 2.3 2.5
[25]:
#
# Método describe aplicado a una columna particular
#
iris.sepal_length.describe()
[25]:
count 150.000000
mean 5.843333
std 0.828066
min 4.300000
25% 5.100000
50% 5.800000
75% 6.400000
max 7.900000
Name: sepal_length, dtype: float64
[26]:
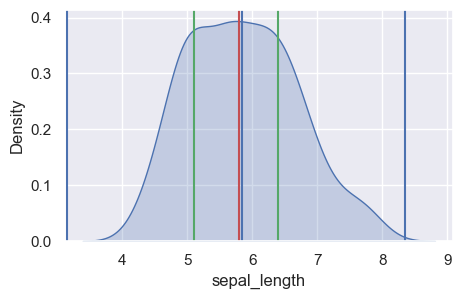
descriptive_stats = iris.sepal_length.describe()
q75 = iris.sepal_length.describe()["75%"]
q25 = iris.sepal_length.describe()["25%"]
iqr = q75 - q25
upper_outliers = q75 + 1.5 * iqr
lower_outliers = q25 - 1.5 * iqr
sns.set(color_codes=True)
plt.figure(figsize=(5, 3))
sns.kdeplot(iris["sepal_length"], shade=True)
plt.axvline(iris.sepal_length.mean())
plt.axvline(iris.sepal_length.median(), color="r")
plt.axvline(q25, color="g")
plt.axvline(q75, color="g")
plt.axvline(upper_outliers, color="b")
plt.axvline(lower_outliers, color="b")
plt.show()
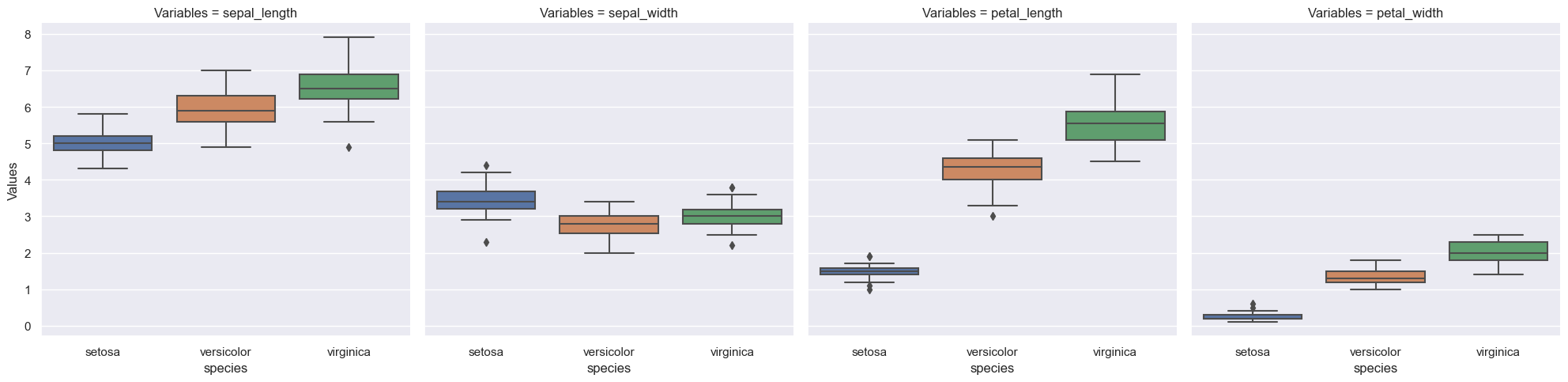
Diagramas de caja y bigote
[27]:
#
# La caja central representa los cuartiles del 25% y 75%.
# La línea centra de la caja representa la mediana
# Los bigotes están ubicados a 1.5 veces el rango interquartil.
# Los puntos por fuera de los bigotes representan outliers.
#
sns.catplot(
x="species",
y="Values",
data=iris_melt,
col="Variables",
kind="box",
col_wrap=4,
)
plt.show()
Medidas de forma
[28]:
from scipy.stats import skew
display(
iris.sepal_length.skew(),
skew(iris.sepal_length),
)
0.3149109566369728
0.3117530585022963
[29]:
from scipy.stats import kurtosis
display(
iris.sepal_length.kurt(),
kurtosis(iris.sepal_length),
)
-0.5520640413156395
-0.5735679489249765