Transformaciones con kernels y kernel trick#
Estas metodologías permiten transformar el espacio de entrada (las características) a un espacio no lineal, con el fin de hacer el problema tratable por las metodologías disponibles.
En el siguiente ejemplo, las dos clases no son linealmente separables. Mediante la transformación a un espacio dimensional mayor:
(x_1, \; x_2) \to (x_1, \; x_2, \; x_1^2 + x_2^2)
las clases se hacen linealmente separables. Note que explicitamente se están computando nuevas columnas en el dataset cuando se agrega la nueva columna x_1^2 + x_2^2.
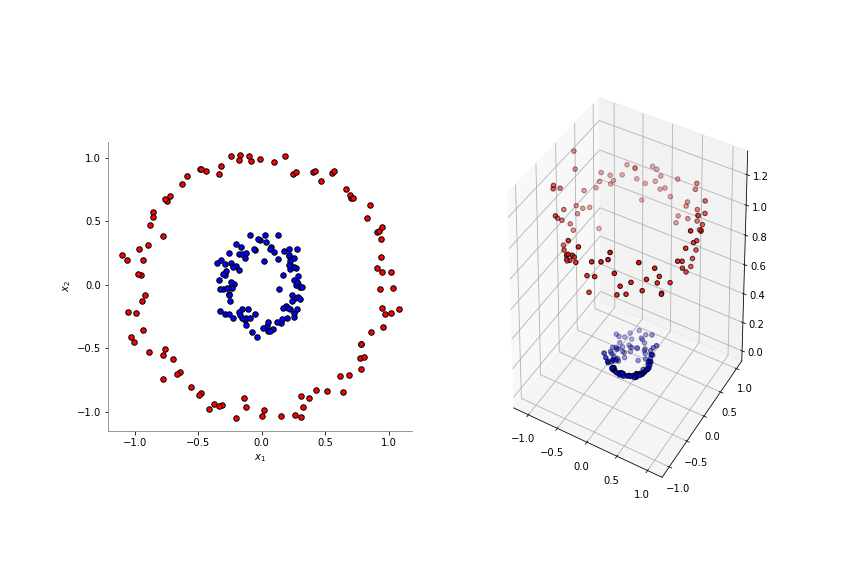
Desde el punto de vista conceptual, existe un número infinito de transformaciones que podrían aumentar la dimensionalidad de los datos, pero muchas de ellas son imprácticas y costosas computacionalmente.
En los métodos de kernel, se soluciona este problema al representar los datos con una matriz K de n \times n (donde n es la cantidad de ejemplos del dataset), donde el elemento k_{ij} corresponde a una comparación de la similitud entre los ejemplos (o patrones) x_i y x_j. Esto se conoce con el nombre de kernel trick.
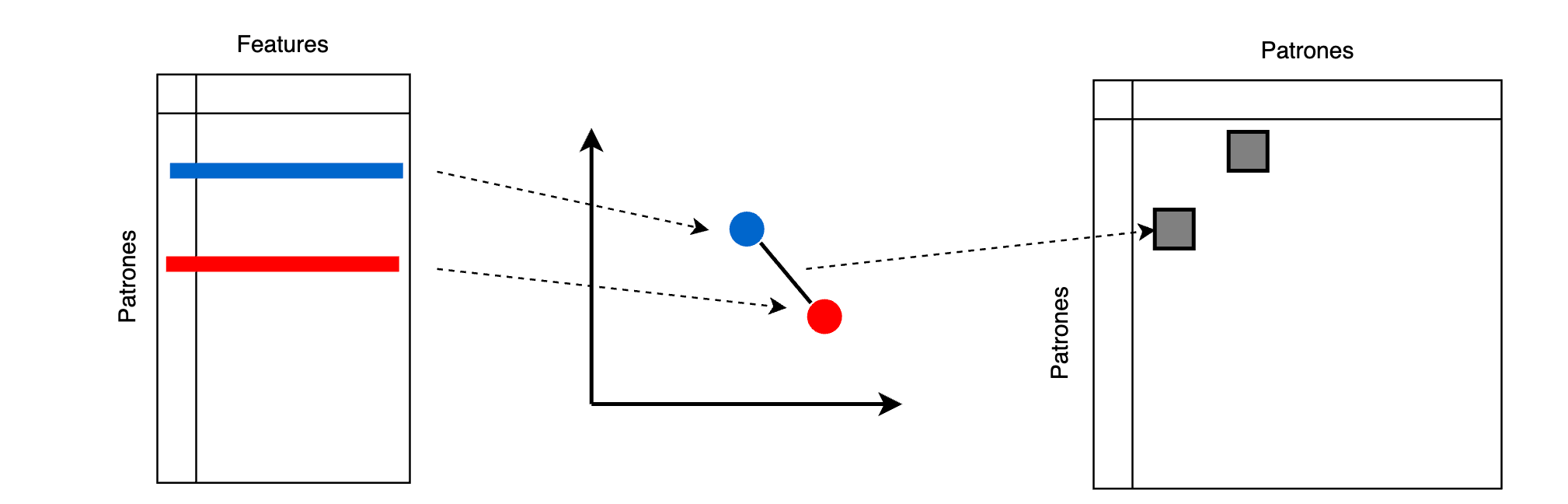
Una función kernel es una función cuyas entradas son los patrones del dataset y retorna su producto punto.
Si \mathbf{x} y \mathbf{z} son dos filas del dataset (patrones) y \phi() es una función, entonces una función kernel se define como:
k(\mathbf{x}, \mathbf{z}) = \phi(\mathbf{x})^T \cdot \phi(\mathbf{z})
Como ejemplo, considere el siguiente kernel lineal:
k(x,y) = x^Ty
para:
[1]:
x = [
[1, 2],
[3, 4],
[5, 6],
]
entonces será igual a:
k(x_1, x_1) = 1 \times 1 + 2 \times 2 = 5
k(x_1, x_2) = 1 \times 3 + 2 \times 4 = 11
y asi sucesivamente.
Kernel Lineal
k(x, y) = x^Ty
[2]:
from sklearn.metrics.pairwise import linear_kernel
linear_kernel(X=x, Y=x)
[2]:
array([[ 5., 11., 17.],
[11., 25., 39.],
[17., 39., 61.]])
[3]:
linear_kernel(X=x)
[3]:
array([[ 5., 11., 17.],
[11., 25., 39.],
[17., 39., 61.]])
Kernel polinomial
k(x, y) = (\gamma x^T y + c_0)^2
[4]:
from sklearn.metrics.pairwise import polynomial_kernel
polynomial_kernel(
X=x,
Y=x,
degree=2,
gamma=1,
coef0=0,
)
[4]:
array([[ 25., 121., 289.],
[ 121., 625., 1521.],
[ 289., 1521., 3721.]])
Kernel sigmoidal
k(x, y) = \tanh(\gamma x^T y + c_0)
[5]:
from sklearn.metrics.pairwise import sigmoid_kernel
sigmoid_kernel(
X=x,
Y=x,
gamma=1,
coef0=0,
)
[5]:
array([[0.9999092, 1. , 1. ],
[1. , 1. , 1. ],
[1. , 1. , 1. ]])
Kernel RBF
k(x, y) = \exp \left(-\gamma ||x - y||^2 \right)
[6]:
from sklearn.metrics.pairwise import rbf_kernel
rbf_kernel(
X=x,
Y=x,
gamma=1,
)
[6]:
array([[1.00000000e+00, 3.35462628e-04, 1.26641655e-14],
[3.35462628e-04, 1.00000000e+00, 3.35462628e-04],
[1.26641655e-14, 3.35462628e-04, 1.00000000e+00]])
Kernel Laplaciano
k(x, y) = \exp \left(-\gamma ||x - y||_1 \right)
donde $ ||x - y||_1$ representa la distancia de Manhattan entre los dos puntos.
[7]:
from sklearn.metrics.pairwise import laplacian_kernel
laplacian_kernel(
X=x,
Y=x,
gamma=1,
)
[7]:
array([[1.00000000e+00, 1.83156389e-02, 3.35462628e-04],
[1.83156389e-02, 1.00000000e+00, 1.83156389e-02],
[3.35462628e-04, 1.83156389e-02, 1.00000000e+00]])
Kernel Chi-cuadrado
k(x, y) = \exp \left( -\gamma \sum_i \frac{(x_i - y_i)^2}{x_i+y_i} \right)
[8]:
from sklearn.metrics.pairwise import chi2_kernel
chi2_kernel(
X=x,
Y=x,
gamma=1,
)
[8]:
array([[1. , 0.1888756 , 0.00940356],
[0.1888756 , 1. , 0.40656966],
[0.00940356, 0.40656966, 1. ]])
[9]:
def fig1():
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets import make_circles
np.random.seed(0)
X, y = make_circles(n_samples=200, factor=0.3, noise=0.05)
plt.figure(figsize=(12, 8))
plt.subplot(1, 2, 1, aspect="equal")
reds = y == 0
blues = y == 1
plt.scatter(X[reds, 0], X[reds, 1], c="red", s=30, edgecolor="k")
plt.scatter(X[blues, 0], X[blues, 1], c="blue", s=30, edgecolor="k")
plt.xlabel("$x_1$")
plt.ylabel("$x_2$")
X_transf = np.c_[X, X[:, 0] ** 2 + X[:, 1] ** 2]
plt.gca().spines["left"].set_color("gray")
plt.gca().spines["bottom"].set_color("gray")
plt.gca().spines["top"].set_visible(False)
plt.gca().spines["right"].set_visible(False)
ax = plt.subplot(1, 2, 2, projection="3d")
ax.scatter(
X_transf[reds, 0],
X_transf[reds, 1],
X_transf[reds, 2],
marker="o",
c="red",
edgecolor="k",
)
ax.scatter(
X_transf[blues, 0],
X_transf[blues, 1],
X_transf[blues, 2],
marker="o",
c="blue",
edgecolor="k",
)
# plt.savefig("assets/kernel_transf.png")
plt.close()
fig1()
